一种利用可用数字数据对 DNA 进行编码的更快方法
一个由分子生物学家、计算机科学家和物理学家组成的国际团队找到了一种将可用数字数据编码到 DNA 链上的方法,速度比目前的方法快 350 倍。在发表于《自然》杂志的研究中,该团队利用 DNA 的表观遗传修饰来创建快速数据存储介质。他们相信,他们的过程可以为利用生物分子系统开发现实世界数据存储开辟新的研究途径。
华盛顿大学的 Carina Imburgia 和 Jeff Nivala 在同一期刊上发表了一篇新闻与观点文章,概述了如何将数据存储在 DNA 链上以及该团队所做的工作。
DNA 本质上是一种存储介质——研究人员估计,一条人类 DNA 链可以存储多达 215,000 TB 的数据。研究团队一直在研究如何将其用作现实世界的数字存储介质。
目前,在使用 DNA 存储大量数字数据之前,有两个主要障碍需要克服。首先是成本。到目前为止,研究人员认为,使用 DNA 作为存储介质的唯一可行方法是在工厂合成,但至少目前而言,这被发现成本太高。
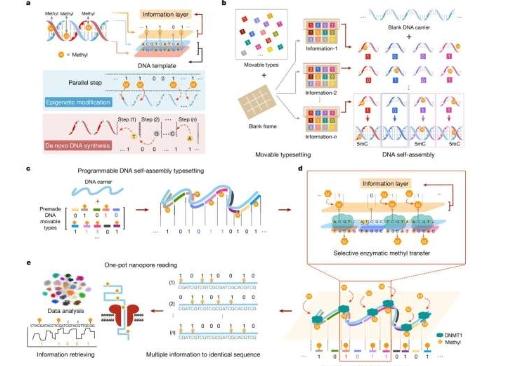
另一个问题是将数据编码到 DNA 链上的速度很慢。在这项新研究中,研究小组找到了一种克服这两个问题的方法,即使用一种自然方法,可以将数据编码速度提高约 350 倍——足以满足实际应用。
这种新方法无需合成,基于通用 DNA 模板中碱基的选择性甲基化作为数据编码手段。研究小组将这种结果描述为 epi- bits,它类似于传统数字计算机使用的位类型。
在这种情况下,1 或 0 表示碱基是否被甲基化。测试表明,它能够以 350 位/反应的速度写入数据,这比之前使用合成 DNA 的方法每次反应写入一位数据有了很大的改进。
免责声明:本答案或内容为用户上传,不代表本网观点。其原创性以及文中陈述文字和内容未经本站证实,对本文以及其中全部或者部分内容、文字的真实性、完整性、及时性本站不作任何保证或承诺,请读者仅作参考,并请自行核实相关内容。 如遇侵权请及时联系本站删除。
